Когда я начинал готовиться к олимпиадам по биологии, часто задавался как выглядят те, или иные белки. Летом прошлого года, нашел-таки как посмотреть 3D визуализацию структур каждого белка у разных организмах. Нельзя передать как я был рад этому. Сколько же часов на просмотр разных белков я потратил… Поэтому, хотел бы поделиться здесь в этими прекрасными ресурсами :3
Вероятно, с этими сайтами уже некоторые знакомы, это национальный центр биотехнологической информаций и SWISS-MODEL центра молекулярных наук о жизни университета Базель.
Ну не давать же просто сайты, и следует дать краткий гайд по использованием сайтами.
-
Находим понравившийся или интересный белок. Для наглядности, хм возьмем гемоглобин
 Ладно, слишком заезженный пример. Возьмем белки: миоглобин человека разумного, и миоглобин гориллы восточной. Далее объясню почему берем сразу два белка.
Ладно, слишком заезженный пример. Возьмем белки: миоглобин человека разумного, и миоглобин гориллы восточной. Далее объясню почему берем сразу два белка. -
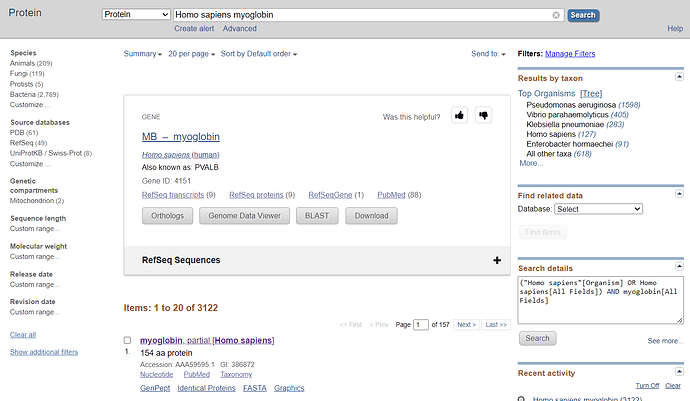
Заходим в ncbi.nlm.nih.gov, и пишем название вида и белка. У нас это Homo Sapiens и Gorilla beringei. Затем myoglobin для каждого запроса. То, что надо подметить следует нажать на protein в databases при запросе, так сайт выдаст FASTA последовательность аминокислот, а не ДНК.
Вводим запрос,
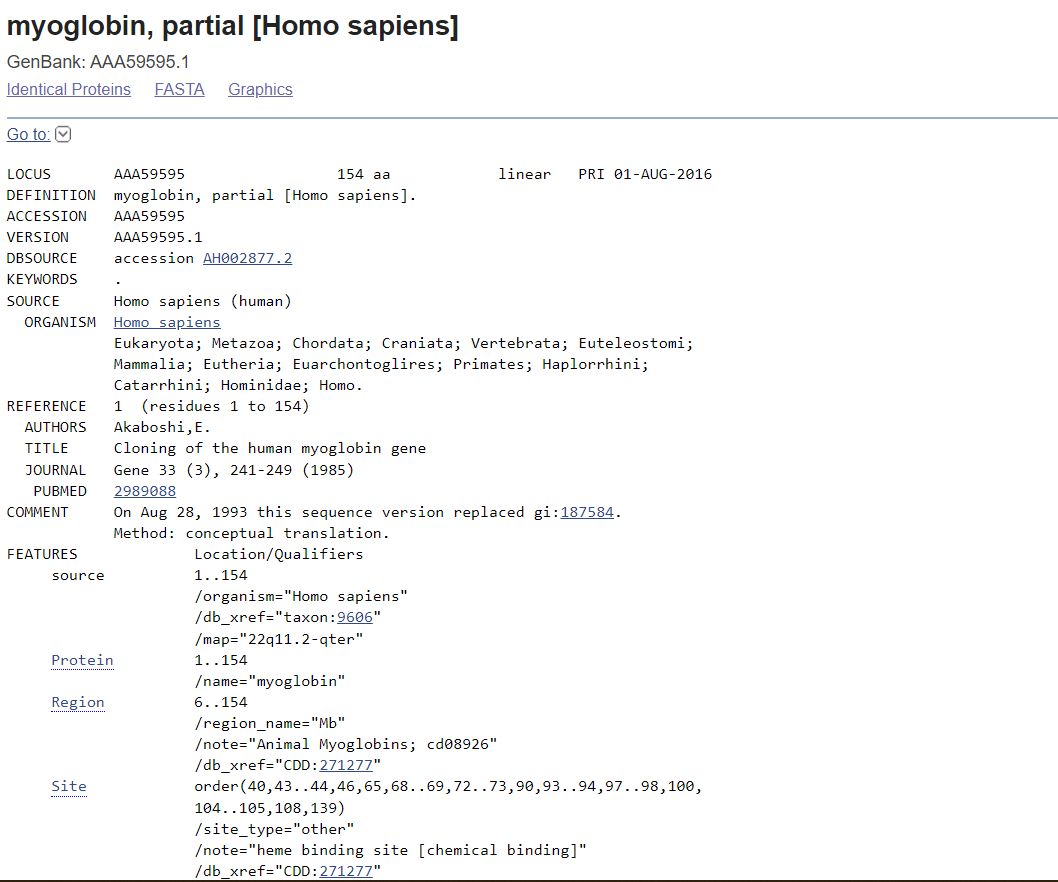
непосредственно переходим на сам белок.
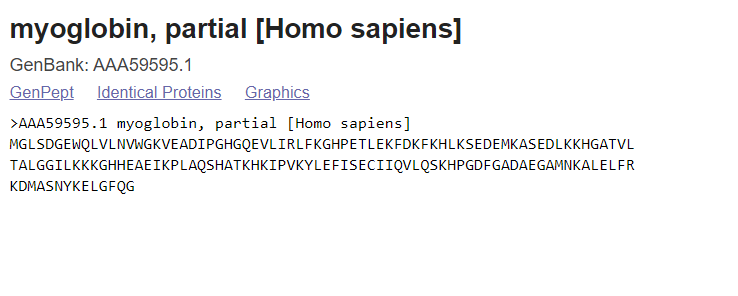
Здесь есть имена авторов, журнал где о нем впервые опубликовали данные и т.п. Но, сейчас нам интересна последовательность аминокислот. Для этого перейдем на FASTA (это текстовый формат последовательности нуклеотидов или аминокислот), и получаем последовательность миоглобина:
Копируем ее для ее дальнейшего использования.
Аналогичные действия повторяем и с миоглобином Gorilla beringei.
FASTA обеих:
>AAA59595.1 myoglobin, partial [Homo sapiens]
MGLSDGEWQLVLNVWGKVEADIPGHGQEVLIRLFKGHPETLEKFDKFKHLKSEDEMKASEDLKKHGATVL
TALGGILKKKGHHEAEIKPLAQSHATKHKIPVKYLEFISECIIQVLQSKHPGDFGADAEGAMNKALELFR
KDMASNYKELGFQG
>sp|P02147.2|MYG_GORBE RecName: Full=Myoglobin
MGLSDGEWQLVLNVWGKVEADISGHGQEVLIRLFKGHPETLEKFDKFKHLKSEDEMKASEDLKKHGATVL
TALGGILKKKGHHEAEIKPLAQSHATKHKIPVKYLEFISECIIQVLQSKHPGDFGADAQGAMNKALELFR
KDMASNYKELGFQG
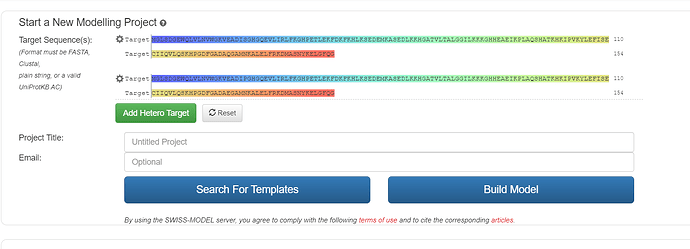
- После вышеуказанных действий заходим на сайт SWISS-MODEL переходим в моделирование. Здесь мы и используем ранее скопированные FASTA тексты. Вставляем сперва человеческое, затем нажмем на add hetero target и туда вставляем последовательность миоглобина гориллы восточной. Мы взяли для обеих видов, чтобы сравнить между собой их структуры (это еще одна функция данного сайта).
Кликаем на build model. Затем ждем. Ждем немало.
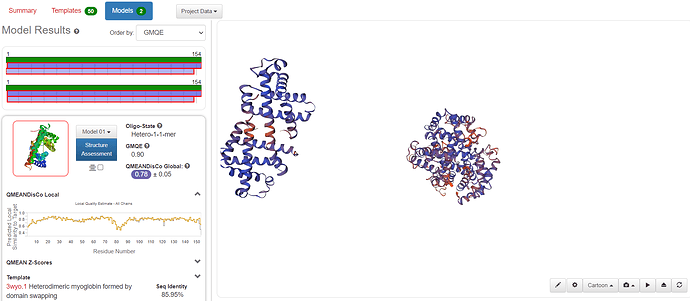
И вот, вышло
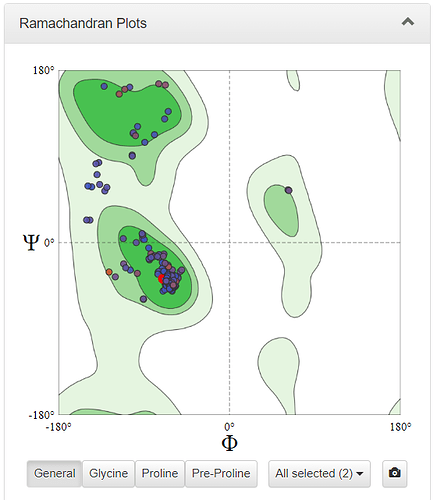
Для отображение обеих белков нужно нажать на их маленькие модели в колонке на правой стороне. Теперь их можно крутить вертеть, “залезать внутрь” и клацать на каждый завиток альфа спирали для просмотра позиций аминокислоты. Потом можно залезать уже в подробности, если перейти на structure assesment. В данном разделе можно увидеть карту Рамачандрана (это градус вращениев N and С terminus каждой аминокислоты), электроотрицательность структур. Можно вдоволь исследовать огромное количество белков и побаловаться с их 3D структурой.
Надеюсь это было вам полезно и познавательно) Эти сайты наверняка не являются уникальными в своем роде, но они довольно широко используются и их владельцы весьма известны в своей сфере, следовательно, у них база данных ооочень огромна.
P.S. Забыл упомянуть довольно интересный момент с библиотекой ncbi. Если введем название белка у довольно хорошо изученного организма (это модельные организмы, вы вряд ли сможете найти у организма, являющиеся менее изученным), и выберем Gene в базе данных, то сможем увидить основную функцию введенного белка, количество экзонов и интронов, расположение его ДНК на хромосоме, уровень экспрессии в тканях и список всевозможных изученной информации будет у вас в руках.
Пример на миоглобине человек вот тут.
Следовательно, можно просто рандомно находить белки и читать их функции и информацию про них. Кто знает может эти знания пригодятся на олимпиаде , и полученные баллы способствуют прохождению на следующий этап ![]()
Автор: Батыр Ержанұлы